Grundlagen der fortgeschrittenen Teilchenphysik-Analyse
Technisches setup
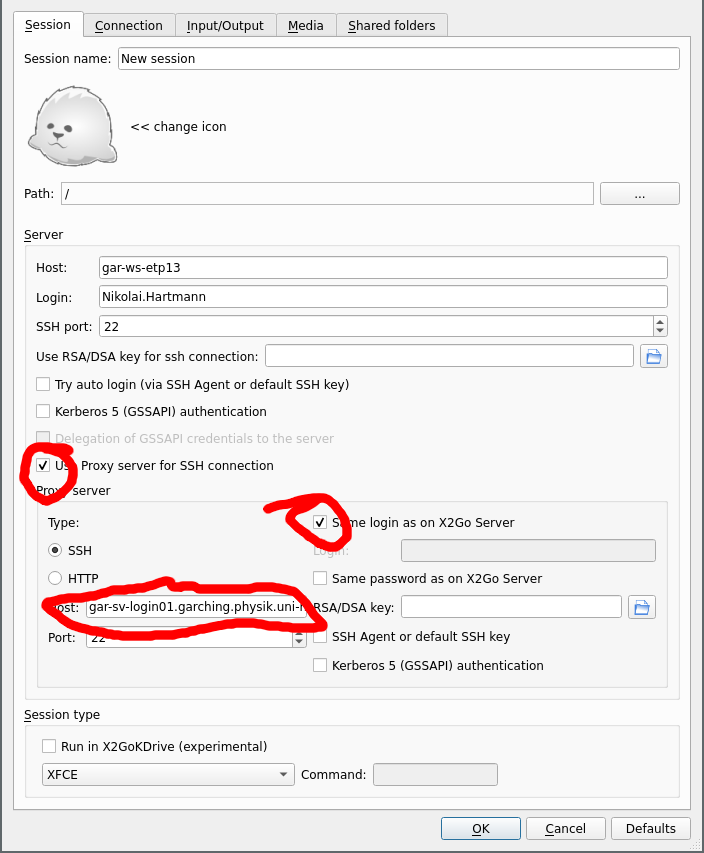
Für dieses Tutorial braucht ihr ssh oder X2GO Zugriff auf die Maschinen in Garching. Falls ihr im VPN seid sollte der Zugriff über <rechner-name>.garching.physik.uni-muenchen.de funktionieren. Ohne VPN kann man über den login server mit -J (ProxyJump) verbinden, z.B. (etpXY durch zutreffendes ersetzen)
ssh -XY -J <Benutzername>@gar-sv-login01.garching.physik.uni-muenchen.de <Benutzername>@gar-ws-etpXY.garching.physik.uni-muenchen.de
Oder entsprechend in ~/.ssh/config konfigurieren
Host gar-ws-etp*
User <Benutzername>
ProxyJump <Benutzername>@gar-sv-login01.garching.physik.uni-muenchen.de
ForwardX11 yes
ForwardX11Trusted yes
Und dann mit ssh gar-ws-etpXY verbinden (2x Passwort eingeben). In X2GO gibt es die Option "Use Proxy Server for SSH connection":
Die typische Teilchenphysik-Analyse
- Als Input: viele rekonstruierte Teilchenkollisionen (GB-TB)
- Metainformationen: Laufende Nummerierung, Trigger
- rekonstruierte Teilchen: Myonen, Elektronen, Jets, MET, ...
- Anwendung von Qualitätskriterien (mittels vorgefertigter Tools)
- Anwendung von Korrekturen auf Physikobjekte (Tools)
- Selektion von gewünschten Ereignissen
- z.B. Ich will nur Ereignisse mit genau 2 Myonen mit \(p_T > 20 GeV\) und genau 2 Jets mit \(p_T > 25 GeV\), die kein b-Meson enthalten und einen Winkel ...
- Histogramme von Energien, Impulsen, Winkeln, Ergebnisse von boosted Decision Trees, Neuronalen Netzen, ...
- Statistische Auswertung
Software Framework I
- Fortgeschrittene ATLAS Datenanalysen werden vorwiegend in C++ geschrieben
- zunehmend auch Python, oder Mischungen verschiedener Technologien
- Alles was >1 Personen benutzen wird früher oder später in Software-Paketen zusammengefasst, welche im Idealfall modular miteinander funktionieren
- Ab einer ausreichenden Menge an Code und Funktionalität kann man von einem Software Framework sprechen, welches
- Software konfiguriert, läd und ausführt
- für die Kommunikation zwischen den Komponenten sorgt
- generelle Funktionalität für verschiedene Aufgaben liefert
- Das liefert eine Umgebung in der geläufige Aufgaben direkt ausgeführt und leicht durch eigenen Code erweitert werden können
Software Framework II
- Ein sehr einfaches Beispiel kennt ihr schon -> Root
- Es gibt eine Vielfalt an Frameworks in ATLAS (und unserem Lehrstuhl) die mehr oder weniger das Gleiche machen
- Wir werden euch hier nur beispielhaft zeigen wie ein Analyse Code funktioniert und zu benutzen ist, die Details erfahrt ihr von euren Betreuern
Wo läuft Analyse-Software
- Das Grid
- mehr als 170 Rechenzentren in 42 Ländern
- Nur für authorisierte CERN Mitglieder
- Batchsysteme
- Mehrere lokale Rechner mit einer gemeinsamen Warteschlange
- SLURM, SGE, condor, ...
- Lokaler Rechner
- Einfach zu benutzen
- Einfach zu debuggen
Generelle Vorgehensweise von Datenanalyse I
- Initialisierung 1x am Anfang
- Dateien öffnen
- Histogramme erzeugen
- Tools initialisieren
- Bearbeitung jedes einzelnes Ereignisses (N mal)
- Anwenden der Tools
- Alle Art von Berechnung
- Füllen der Histogramme
- Finalisierung 1x am Ende
- Aufräumen
- Ergebnisse speichern
Prinzipielle Idee der meisten Frameworks
Generelle Vorgehensweise von Datenanalyse II
bool Analysis::initialize(){
...
std::cout << "Number of total events: " << n_events << std::endl;
...
return true;
}
bool Analysis::execute(){
...
my_histogram->Fill(muon_pt);
...
return true;
}
bool Analysis::finalize(){
...
my_histogram->Write();
...
return true;
}
NTuples
- "Rohes" Root Datenformat, ähnlich Excel-Tabelle
- Jede "Spalte" enthält Zahlen oder Listen von Zahlen (oder Listen von Listen von Zahlen)
- Häufig letzter Schritt in einer Analyse (klein genug um auf einem einzelnen Rechner verarbeitet zu werden)
- Einfach mit ROOT benutzbar (z.B. via
TTree::Draw)
- (Inzwischen) einfach in andere Formate konvertierbar (z.B pandas Dataframe)
- Bei komplizierteren Daten viel Buchhaltung notwendig ("Index-Schlacht"), häufig wird hierfür eine übergeordnete Klassenstruktur in Frameworks benutzt
Beispiel: Um N Myonen eines Events zu beschreiben sind muon_px, muon_py, muon_pz, muon_E Listen von je N doubles.
muon_py[3] // ist die Impulskomponente in y-Richtung des 4. Myons.
Tipps und Tricks zu scripts mit TTree::Draw
Dokumentation
TTree::Draw unterstützt eine ganze Reihe von Ausdrücken. Die Dokumentation im ROOT Reference Guide ist relativ ausführlich
https://root.cern/doc/master/classTTree.html#a73450649dc6e54b5b94516c468523e45
TDirectory
Um ein Histogramm mit TTree::Draw zu füllen muss es dem gleichen TDirectory zugeordnet sein wie der TTree. Das geschieht automatisch wenn das Histogramm erstellt wird nachdem ein TFile geöffnet wurde
rootfile = ROOT.TFile.Open(filename)
tree = rootfile.Get(treename)
hist = ROOT.TH1F(histname, title, nbins, xmin, xmax)
Nach dem Befüllen kann dann das assoziierte Directory auf 0 gesetzt werden, sofern das Histogramm nicht in einer ROOT Datei gespeichert werden soll. Andernfalls wird das Histogramm gelöscht wenn TFile::Close aufgerufen wird!
Nicht-interaktiv
Um TTree::Draw nicht-interaktiv zu verwenden (kein erstellen von TCanvas beim Aufruf) kann TTree::Project oder die option "goff" verwendet werden
tree.Draw("{}>>{}".format(expr, histname), cut, "goff")
Gewichtete Ereignisse
TTree::Draw akzeptiert ein Argument cut mit dem zugleich eine Selektion und auch eine Gewichtung mitgegeben werden kann (Ereignisse mit Gewicht 0 füllen das Histogramm nicht).
tree.Draw("lep_pt[0]>>myhist", "(lep_n<2)*(mcWeight)", "goff")
Der Standardabweichung einer gewichteten Summe einer poissonverteilten Anzahl Ereignisse lässt sich über \(\sqrt{\sum w_i^2}\) abschätzen. Damit ROOT diese Summe der quadrierten Gewichte pro Bin speichert wird vor dem Füllen des Histogramms folgender Aufruf benötigt
Kleine Übung: TTree::Draw in einem python script
Ein kleines Beispiel:
cp /project/etp3/bachelorkurs2019/fill_hist.py .
./fill_hist.py
evince plot.pdf
Für kompliziertere Plots könnt ihr in der Regel auf fertigen Code zurückgreifen der für die jeweiligen Analysen entwickelt wurde (bekommt ihr von euren Betreuern). Ein etwas allgemeineres Framework für einige "Standardplots" mit Ntuples ist MPF:
Code
Dokumentation
Beispiele von xAOD Datentypen
xAOD::MuonContainer *muons // Liste aller Myonen im Event
xAOD::Muon *mu = muon->at(0); // einzelnes Myon, gepickt aus der Liste
xAOD::ElectronContainer, xAOD::Electron, xAOD::JetContainer, xAOD::Jet, ...
xAOD::EventInfo // Metainformationen über das Event, ob Simulation oder Daten, Eventnummer, ...
Eine volle Liste aller Objekte in einem xAOD gibt checkxAOD.py (in der später aufgesetzten Umgebung)
checkxAOD.py /project/etp3/bachelorkurs2019/xAODs/mc16_13TeV_Wmunu/DAOD_SUSY5.16135178._000014.pool.root.1
ATLAS Software Umgebung
- Hier am Lehrstuhl: Ubuntu Linux
- Alle ATLAS Software kompiliert für Scientific Linux/CentOS (offizielle CERN Linux Distribution)
- Inkompatibilitäten -> Wechsel zu einheitlichem System mittels Singularity
Nach Eingabe von
source /project/etpsw/Common/bin/setup_image.sh
kann man mit folgendem Befehl in eine CentOS 7 Umgebung wechseln
In dieser kann man dann die ATLAS Software Umgebung von CVMFS aufsetzen
export ATLAS_LOCAL_ROOT_BASE=/cvmfs/atlas.cern.ch/repo/ATLASLocalRootBase
alias setupATLAS='source ${ATLAS_LOCAL_ROOT_BASE}/user/atlasLocalSetup.sh'
setupATLAS
Um sich das Leben einfacher zu machen kann man sich diese 3 Zeilen auch in "~/.bashrc" schreiben
source /project/etpsw/Common/bin/setup_image.sh
export ATLAS_LOCAL_ROOT_BASE=/cvmfs/atlas.cern.ch/repo/ATLASLocalRootBase
alias setupATLAS='source ${ATLAS_LOCAL_ROOT_BASE}/user/atlasLocalSetup.sh'
dann hat man centos7 und setupATLAS in jeder neuen Konsole zur Verfügung.
CVMFS
Netzwerk-Dateisystem, das unsere spezielle ATLAS-Software bereitstellt (ROOT, Athena, ...)
Ausführen des Befehls auf der vorherigen Folie:
centos7 # (falls noch nicht in einer CentOS Umgebung)
setupATLAS
Zeigt eine Übersicht zum Aufsetzen einzelner Pakete an.
Zum Beispiel um ROOT aufzusetzen
ACM (asetup und CMake)
- Tool um Analysen zu verwalten und kompilieren
- Setzt die Software-Umgebung auf, in der ATLAS Analysen für xAODs ausgeführt werden können (AnalysisBase)
Aufsetzen einer bestimmten Version von AnalysisBase
acmSetup AnalysisBase,21.2.116
Umgebung wieder-aufsetzen (im "build" Verzeichnis, siehe nächste Seite)
Alles Kompilierte löschen, sauberer Neuanfang
Lokale Pakete finden
Kompilieren
Kleine xAOD Analyse: Umgebung aufsetzen
# In ein temporäres verzeichnis wechseln (nur für dieses Tutorial)
TEMPDIR=$(mktemp -d /tmp/$(id -un)_XXXX)
cd $TEMPDIR
# Sofern nicht in "~/.bashrc"
source /project/etpsw/Common/bin/setup_image.sh
# Falls noch nicht in centos7 Umgebung
centos7
# Sofern nicht in "~/.bashrc"
export ATLAS_LOCAL_ROOT_BASE=/cvmfs/atlas.cern.ch/repo/ATLASLocalRootBase
alias setupATLAS='source ${ATLAS_LOCAL_ROOT_BASE}/user/atlasLocalSetup.sh'
# ATLAS Software von CVMFS aufsetzen
setupATLAS
# Analyse Skelett herkopieren und in build Verzeichnis wechseln
cp -r /project/etp3/bachelorkurs2020/ROOTAnalysisTutorial .
cd ROOTAnalysisTutorial/build
# AnalysisBase aufsetzen und CMake ausführen
acmSetup AnalysisBase,21.2.116
Wenn alles gut gegangen ist, sollte erscheinen:
ACM Setup complete. Use acm help to get started.
Erste Kompilierung
In der selben Konsole
Erfolg wenn letzte Zeile lautet
[100%] Built target Package_MyAnalysis
Code ausführen mittels (in einem dediziertem "run" Verzeichnis)
cd ../run
runAnalysis.py /project/etp3/bachelorkurs2020/xAODs/mc16_13TeV_Wmunu/DAOD_SUSY5.16135178._000014.pool.root.1
Ergebnisse werden in submitDir gespeichert
Dieses Verzeichnis wird bei jedem Ausführen von runAnalysis.py überschrieben. Sofern Ergebnisse behalten werden sollen müssen wir sie woanders hin tun:
mv submitDir results-data
Intermezzo: Text editieren
Falls ihr mit x2go eingeloggt seid (oder lokal angemeldet) könnt ihr in in einer NEUEN Konsole (die CentOS Umgebung hat relativ wenig extra Programme) einen Texteditor (z.B. kate) öffnen
# temporäres Verzeichnis suchen
ls -d /tmp/$(id -un)*
# Verzeichnis betreten
cd <pfad-zu-temporärem-verzeichnis>/ROOTAnalysisTutorial/source
# Dateien mit kate öffnen
kate MyAnalysis/Root/MyxAODAnalysis.cxx \
MyAnalysis/MyAnalysis/MyxAODAnalysis.h \
MyAnalysis/CMakeLists.txt \
MyAnalysis/share/runAnalysis.py
Alternative 1: sshfs
Falls ihr auf Linux oder macOS arbeitet und dort bereits einen Texteditor installiert habt könnt ihr mit Hilfe von sshfs (sollte in den meisten Distributionen mit dem Paketmanager installierbar sein) das Arbeitsverzeichnis auf eurem lokalen Rechner mounten und die Dateien mit dem lokalen Texteditor editieren. Z.B.
# (das lokale verzeichnis sollte leer sein)
sshfs -o ssh_command="ssh -J <Benutzername>@gar-sv-login01.garching.physik.uni-muenchen.de" \
<Benutzername>@<remote-workstation>:<pfad-zu-remote-verzeichnis> <pfad-zu-lokalem-verzeichnis>
Das lokale Verzeichnis sollte leer sein. Nach dem Aufruf von sshfs solltet ihr darin den Inhalt des Remote Verzeichnisses sehen können. remote-workstation ist der Name eures Arbeitsrechners z.B. gar-ws-etp01. Falls ihr direkt auf dem login server arbeitet (nicht dauerhaft zu empfehlen, aber fürs Tutorial ok) oder ein Verzeichnis mounten wollt dass auf dem login server verfügbar ist könnt ihr direkt über den login server mounten:
sshfs <Benutzername>@gar-sv-login01.garching.physik.uni-muenchen.de:<pfad-zu-remote-verzeichnis> <pfad-zu-lokalem-verzeichnis>
Alternative 2: vim oder andere textbasierte Editoren
Falls ihr mit vim vertraut seid könnt ihr diesen Editor auch direkt in der ssh Konsole ausführen (ist sowohl auf dem Rechner als auch in der centos7 Umgebung installiert). Alternative gibt es (in der Ubuntu Umbgebung) emacs und nano.
Überblick über die Dateien
- MyAnalysis/share/runAnalysis.py: Steuerprogramm. Setzt Eventloop spezifische Einstellungen auf und führt MyxAODAnalysis aus.
- MyAnalysis/MyAnalysis/MyxAODAnalysis.h: Headerdatei. Hier Paket-Header-Dateien inkludieren, Histogramme und Counter deklarieren.
- MyAnalysis/Root/MyxAODAnalysis.cxx: Hauptdatei, analog TSelector.C.
- MyAnalysis/CMakeLists.txt: CMake Konfigurationsdatei. Steuert die Kompilierung.
Daten lesen
Wir werden uns hier auf Myonen beschränken, da diese ohne viel Aufwand eine saubere Signatur liefern
Für Myonen wird das Paket xAODMuon benötigt, d.h. wir müssen es in MyAnalysis/CMakeLists.txt nach LINK_LIBRARIES AnaAlgorithmLib hinzufügen:
LINK_LIBRARIES AnaAlgorithmLib xAODMuon
und in MyAnalysis/MyxAODAnalysis.cxx das folgende Include Statement am Anfang der Datei:
#include "xAODMuon/MuonContainer.h"
In MyAnalysis/MyxAODAnalysis.cxx, Funktion StatusCode MyxAODAnalysis :: execute ():
const xAOD::MuonContainer* muons = 0;
evtStore()->retrieve( muons, "Muons" );
Info("execute", "Anzahl Myonen: %ld", muons->size());
for (const xAOD::Muon* muon: *muons) {
// Grössen wie Energie und Impuls werden konventionell in MeV gespeichert -> Umrechnung in GeV
Info("execute()", " Originaler Myon pt = %.3f GeV", (muon->pt() * 0.001));
}
Warnung: Info() Funktion erzeugt hierbei SEHR viel Output, nur für Testzwecke sinnvoll!
Nach jeder Änderung am Code ist es notwendig erneut zu kompilieren:
acm compile
runAnalysis.py /project/etp3/bachelorkurs2020/xAODs/mc16_13TeV_Wmunu/DAOD_SUSY5.16135178._000014.pool.root.1
# Abbrechen mit Strg+c
Histogramme
Eventloop übernimmt einen Grossteil des Output Managements, das einzige was zu tun ist ist am Anfang von MyxAODAnalysis.cxx den ROOT Histogram Header zu inkludieren:
Wir können nun Gebrauch von der book und hist Funktion in Eventloop machen um Histogramme zu initialisieren und zu befüllen. Um z.B. ein Histogramm für den Myon Impuls zu erzeugen müssen wir book in initialize () aufrufen.
In MyAnalysis/MyxAODAnalysis.cxx, Funktion StatusCode MyxAODAnalysis :: initialize ():
book(TH1F("h_muon_pt", "Transverser Myon Impuls", 100, 0, 100));
In der Schleife über die Myonen können wir nun das Histogramm befüllen, welches wir mit hist aufrufen mit dem Namen "h_muon_pt" den wir bei book vorher benutzt haben.
In MyAnalysis/MyxAODAnalysis.cxx, Funktion StatusCode MyxAODAnalysis :: execute ():
for (const xAOD::Muon* muon: *muons) {
// Grössen wie Energie und Impuls werden konventionell in MeV gespeichert -> Umrechnung in GeV
Info("execute()", " Originaler Myon pt = %.3f GeV", (muon->pt() * 0.001));
// Histogram befüllen
hist("h_muon_pt")->Fill(muon->pt() * 0.001 );
}
Kompilieren und Code ausführen:
acm compile
runAnalysis.py /project/etp3/bachelorkurs2019/xAODs/mc16_13TeV_Wmunu/DAOD_SUSY5.16135178._000014.pool.root.1
Jetzt sollte das Histogramm in dem Output submitDir/hist-xAOD.root sein. Mit ROOT öffnen, und mit TBrowser ansehen:
rootbrowse submitDir/hist-xAOD.root
Physik!
Interessante Messgrössen für Ereignisse mit Zwei Myonen sind unter Anderem die invariante Masse des Zwei-Myon-Systems und der Öffnungswinkel zwischen den Myonen.
In MyAnalysis/MyxAODAnalysis.cxx, Funktion StatusCode MyxAODAnalysis :: execute ():
if (muons->size() == 2) { // falls wir genau 2 Myonen haben
const xAOD::Muon *mu1 = muons->at(0); // Numerierung fängt bei 0 an!
const xAOD::Muon *mu2 = muons->at(1);
TLorentzVector dimuon = mu1->p4() + mu2->p4();
double Mll = dimuon.M() *0.001; // umrechnen von MeV in GeV
Info("execute()", "Die Dimuon-Masse ist %.2f GeV", Mll);
double dphi = fabs(mu1->p4().DeltaPhi(mu2->p4()));
Info("execute()", "Der Öffnungswinkel ist %.2f Rad", dphi);
}
Aufgaben
- Erzeugt und füllt Histogramme mit der invarianten Zwei-Myon-Masse und dem Öffnungswinkel
- Optional für Fortgeschrittene: vielleicht auch ein 2D Histogramm aus beiden Grössen (TH2F)
Wie sieht das mit simulierten Daten aus:
runAnalysis.py /project/etp3/bachelorkurs2020/xAODs/mc16_13TeV_Zmumu/DAOD_HIGG3D1.15802786._001411.pool.root.1
Wie sieht es aus wenn wir ein paar mehr echte Daten-Ereignisse hinzu nehmen:
runAnalysis.py /project/etp3/bachelorkurs2020/xAODs/data17_13TeV/*
Was passiert wenn man z.B. verlangt dass der Transversalimpuls der Myonen mindestens 15 GeV betragen muss:
if(mu1->pt() > 15000. && mu2->pt() > 15000.){
// Befülle Histogramme nur wenn Myonen Transversalimpuls mindestens 15 GeV beträgt ("h_mll" und "h_dphi" müssen vorher erzeugt werden)
hist("h_mll")->Fill(Mll);
hist("h_dphi")->Fill(dphi);
}
Optional für Fortgeschrittene: Was ändert sich wenn die Myonen kalibriert werden (nächsten Folien)?
Eine mögliche Lösung findet ihr unter /project/etp3/bachelorkurs2020/ROOTAnalysisTutorial_solution
Event-Typ: Daten oder Simulation
- Wir wollen simulierte Daten (Monte Carlo, MC) mit echten ATLAS-Daten vergleichen
- Programm muss wissen ob Daten MC sind oder nicht, z.B. für Myonenkalibrierung
- EventInfo Objekt hilft weiter, aus dem Paket xAODEventInfo
In MyAnalysis/CMakeLists.txt:
LINK_LIBRARIES AnaAlgorithmLib xAODMuon xAODEventInfo
In MyAnalysis/MyxAODAnalysis.cxx:
#include "xAODEventInfo/EventInfo.h"
In MyAnalysis/MyxAODAnalysis.cxx, Funktion StatusCode MyxAODAnalysis :: execute () (am besten vor der Myonenschleife):
bool isMC = false; // wir nehmen an es ist keine Simulation
const xAOD::EventInfo* eventInfo = 0;
evtStore()->retrieve( eventInfo, "EventInfo"); // wir laden das EventInfo Objekt
if(eventInfo->eventType( xAOD::EventInfo::IS_SIMULATION ) ){
isMC = true; // falls es doch Simulation ist, setzen wir isMC auf wahr
}
Myon Kalibrierung
In simulierten Daten müssen Myonen kalibriert werden, wofür wir das offizielle ATLAS Tool MuonCalibrationAndSmearingTool benutzen. Dazu benutzen wir die Pakete AsgAnalysisInterfaces und MuonAnalysisInterfacesLib.
In MyAnalysis/CMakeLists.txt:
LINK_LIBRARIES AnaAlgorithmLib xAODMuon xAODEventInfo AsgAnalysisInterfaces MuonAnalysisInterfacesLib
In MyAnalysis/MyxAODAnalysis.h:
#include <AsgTools/AnaToolHandle.h>
#include <MuonAnalysisInterfaces/IMuonCalibrationAndSmearingTool.h>
ToolHandle deklarieren in MyAnalysis/MyxAODAnalysis.h, unter public::
// MuonCalibrationAndSmearing
asg::AnaToolHandle<CP::IMuonCalibrationAndSmearingTool> m_muonCalibrationAndSmearingTool; //!
Hinweis: //! ist aus technischen Gründen notwendig!
Das Tool selber soll erst erzeugt werden wenn der Algorithmus erstellt wird. Also schreiben wir eine entsprechende Zeile in die Initializer Liste des Konstruktors in MyAnalysis/MyxAODAnalysis.cxx (am Anfang der Datei, unter den Include Statements). Dieser sollte hinterher so aussehen:
MyxAODAnalysis :: MyxAODAnalysis (const std::string& name, ISvcLocator *pSvcLocator) :
EL::AnaAlgorithm (name, pSvcLocator),
m_muonCalibrationAndSmearingTool ("CP::MuonCalibrationAndSmearingTool/MuonCorrectionTool",this)
{
// Here you put any code for the base initialization of variables,
// e.g. initialize all pointers to 0. Note that things like resetting
// statistics variables should rather go into the initialize() function.
}
In MyAnalysis/MyxAODAnalysis.cxx, Funktion StatusCode MyxAODAnalysis :: initialize () (direkt unterhalb der Histogram Initialisierung):
m_muonCalibrationAndSmearingTool.initialize();
Wir brauchen eine veränderbare Kopie unserer Myonen ("Shallow Copy"). Danach laufen wir über die Myonen und wenden die Kalibrierung an, sofern es sich um MC Ereignisse handelt.
In MyAnalysis/MyxAODAnalysis.cxx:
#include <xAODCore/ShallowAuxContainer.h>
#include <xAODCore/ShallowCopy.h>
In MyAnalysis/MyxAODAnalysis.cxx, Funktion StatusCode MyxAODAnalysis :: execute ():
// Eine "shallow copy" vom Myon Container erstellen
auto muons_shallowCopy = xAOD::shallowCopyContainer( *muons );
std::unique_ptr<xAOD::MuonContainer> muonsSC (muons_shallowCopy.first);
std::unique_ptr<xAOD::ShallowAuxContainer> muonsAuxSC (muons_shallowCopy.second);
// Über die Kopien iterieren
for (xAOD::Muon* muonSC : *muonsSC) {
if (isMC) {
// Kalibrierung anwenden
m_muonCalibrationAndSmearingTool->applyCorrection(*muonSC);
Info("execute()", " Korrigierter Myon pt = %.5f GeV", (muonSC->pt() * 0.001));
}
}
Kompilieren und ausführen:
acm compile
runAnalysis.py /project/etp3/bachelorkurs2019/xAODs/mc16_13TeV_Zmumu/DAOD_HIGG3D1.15802786._001411.pool.root.1